Fingerpieks => 3 Tropfen Blut => gesamte HCV-Diagnostik (Serologie, RNA und Genotyp)
 Schwierige Venenverhältnisse können einer adäquaten Abklärung von i.v.-Drogenkonsumenten bezüglich Hepatitis C im Wege stehen. HCV-Antikörper, -RNA- und -Genotyp-Bestimmung sind neu schon mit nur 3 Tropfen (150ul) auf Filterpapier getrocknetem kapillären Vollblut möglich.
Schwierige Venenverhältnisse können einer adäquaten Abklärung von i.v.-Drogenkonsumenten bezüglich Hepatitis C im Wege stehen. HCV-Antikörper, -RNA- und -Genotyp-Bestimmung sind neu schon mit nur 3 Tropfen (150ul) auf Filterpapier getrocknetem kapillären Vollblut möglich.
Was bei HIV – vor allem in 3. Welt-Ländern – längst gut etabliert ist, nämlich die HIV-Antikörper- und -RNA-Bestimmung in auf Filterpapier getrockneten Blutstropfen (DBS = dried blood spot), steckte für die HCV-Diagnostik bislang noch in den Kinderschuhen. Nun hat eine diesen Monat in Hepatology publizierte Französische Studie gezeigt, dass mit der DBS-Methode unter Verwendung kommerziell erhältlicher serologischer bzw. molekularer Assays der Nachweis von HCV-Antikörpern sowie die Quantifizierung und Genotypisierung von HCV-RNA zuverlässig möglich sind.
Methoden:
Von insgesamt 200 Patienten (100 anti-HCV-negativ, 100 anti-HCV-positiv => davon 62 mit nachweisbarer HCV-RNA) wurden gepaarte Serum- und DBS-Proben abgenommen. Die DBS-Proben bestanden aus drei Tropfen à 50ul Vollblut, welche auf drei 12mm durchmessende, vorgedruckte Kreise einer Filterpapierkarte (Whatman 903 card) aufgetragen wurden. Nach 18stündiger Trocknung bei Raumtemperatur wurden die Filterpapierkarten einzeln in verschliessbare Plastiksäckchen abgepackt und innert 48 Stunden nach Blutentnahme bei -20°C tiefgefroren. Die Analyse der DBS-Proben erfolgte 1-8 Wochen später.

Für den anti-HCV-Test wurde eine 6mm durchmessende Scheibe aus der DBS-Probe auf dem Filterpapier herausgestanzt, in ein Well einer unbeschichtete Microtiter-Platte überführt und mit 200ul Probenverdünnung versetzt über Nacht unter sanftem Schütteln inkubiert. Am anderen Tag erfolgte die anti-HCV-Bestimmung mittels Ortho HCV 3.0 ELISA (enzyme-linked immunosorbent assay), wobei anstelle von 20ul Serum + 200ul Probenverdünnung 100ul des DBS-Eluats verwendet wurden. Die verbleibenden 100ul DBS-Eluat setzte man bei 20 ausgewählten Proben zur Durchführung eines Immunoblots (INNO-LIA HCV Score) als Bestätigungstest ein.
Für die HCV-RNA-Extraktion wurden zwei 6mm durchmessende Scheiben aus der DBS-Probe auf dem Filterpapier herausgestanzt, in 1,5ml-Eppendorf-Hütchen überführt, mit 400ul Pufferlösung versetzt und unter kontinuierlichem Schütteln bei 4°C während ca. 2 Stunden inkubiert. Nach dem Abzentrifugieren erfolgte die RNA-Extraktion aus dem Überstand mit dem automatisierten Cobas Ampliprep Total Nucleic Acid Isolation 100 kit. Zur HCV-RNA-Quantifizierung verwendete man den Cobas TaqMan HCV test mit einer unteren Nachweisgrenze von 15 U/ml.
Resultate:
1) Probenstabilität
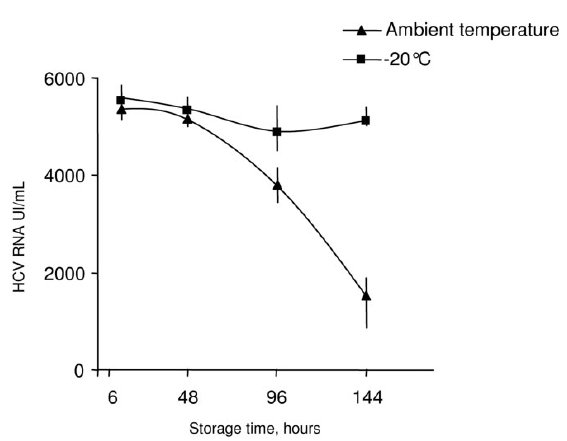 Für eine optimale, reproduzierbare HCV-RNA-Ausbeute aus dem Whatman 903-Papier muss die DBS-Probe innert 48 Stunden nach Blutentnahme und Trocknung bei -20°C tiefgefroren werden. Eine verlängerte Lagerung bei Raumtemperatur führte zu falsch tiefen HCV-RNA-Werten. Demgegenüber nahm die Rate falsch positiver HCV-Antikörpertest mit der Dauer der Raumtemperatur-Exposition zu (nach 6 Tagen 4/5 anti-HCV-Negative falsch positiv), während sämtliche initial HCV-positive DBS-Proben positiv blieben. (vgl. Abbildungen, oben: HCV-RNA; unten: ELISA, gestrichelte Linie = cutoff)
Für eine optimale, reproduzierbare HCV-RNA-Ausbeute aus dem Whatman 903-Papier muss die DBS-Probe innert 48 Stunden nach Blutentnahme und Trocknung bei -20°C tiefgefroren werden. Eine verlängerte Lagerung bei Raumtemperatur führte zu falsch tiefen HCV-RNA-Werten. Demgegenüber nahm die Rate falsch positiver HCV-Antikörpertest mit der Dauer der Raumtemperatur-Exposition zu (nach 6 Tagen 4/5 anti-HCV-Negative falsch positiv), während sämtliche initial HCV-positive DBS-Proben positiv blieben. (vgl. Abbildungen, oben: HCV-RNA; unten: ELISA, gestrichelte Linie = cutoff)
(96h=4d, 144h=6d, 288h=12d)
2) HCV-Antikörper-Test
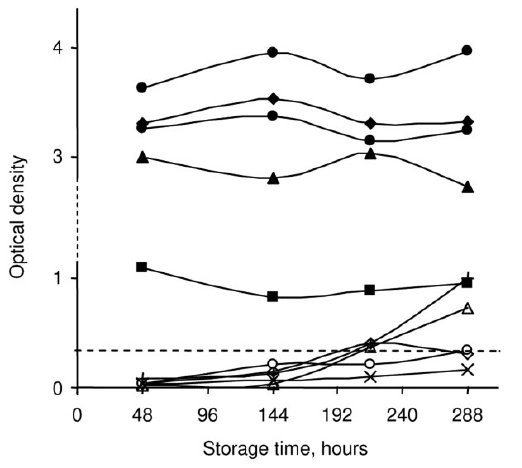
 Bei einem Cuttoff-Wert von 0,380 (optical density = OD) hatte der verwendete HCV-ELISA-Test in den DBS-Proben eine Sensitivität von 99% und eine Spezifität von 98% (OD für HCV-Positive 2,76+/-1,06; OD für HCV-Negative 0,057+/-0,053).
Bei einem Cuttoff-Wert von 0,380 (optical density = OD) hatte der verwendete HCV-ELISA-Test in den DBS-Proben eine Sensitivität von 99% und eine Spezifität von 98% (OD für HCV-Positive 2,76+/-1,06; OD für HCV-Negative 0,057+/-0,053).
Eine DBS-Probe von einem HIV-positiven Patienten lieferte mit einer OD von 0,112 ein falsch-negatives Resultat. Auch im Serum war anti-HCV nur schwach positiv (Immunoblot positiv, HCV-RNA nicht nachweisbar). Ein anderer Patient, welcher mit einer OD von 0,404 nur knapp oberhalb des Cuttoffs lag, war im Serum anti-HCV-negativ und zeigte einen negativen HCV-Immunoblot, so dass von einem falsch-positiven Resultat auszugehen ist. (vgl. Abbildung)
Zur Ermittlung der unteren Nachweisgrenze wurden sieben anti-HCV-positive DBS- und Serumproben mit HCV-negativem Blut bzw. Serum verdünnt. Die untere Nachweisgrenze lag für die DBS-Proben im Median bei einem Verdünnungstiter von 1/512 (Range: 1/64-1/4096) und für die Serumproben bei einem medianen Verdünnungstiter von 1/1024 (Range: 1/512-1/65.336), also 2-3 Verdünnungsstufen höher als in der dazugehörigen DBS-Probe.
In 10 anti-HCV-positiven und 10 anti-HCV-negativen DBS-Proben sowie den dazugehörigen Serumproben wurde zur Bestätigung ein HCV-Immunoblot durchgeführt. Es zeigte sich eine 100%ige Konkordanz (keine falsch-positiven, falsch-negativen oder indeterminanten Resultate).
3) quantitative HCV-RNA-Bestimmung
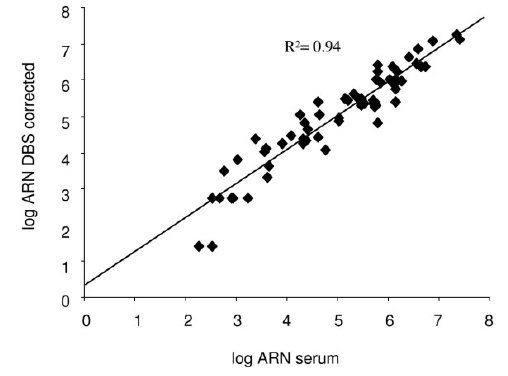
 Für 62 Patienten mit chronischer Hepatitits C wurden die HCV-RNA-Werte in den DBS-Proben mit denen in den gepaarten Serum-Proben verglichen. In 60/62 (97%) der DBS-Proben war HCV-RNA nachweisbar. Im entsprechenden Serum der zwei HCV-RNA-negativen DBS-Proben wurde eine niedrige Viruslast von nur 178 bzw. 331 U/ml gemessen. Fünf DBS-Proben waren zwar HCV-RNA-positiv, jedoch mit Werten unterhalb der Nachweisgrenze von 15 U/ml (HCV-RNA in den dazugehörigen Serumproben: 334 bis 1672 U/ml). Somit konnte in 55/62 (89%) der DBS-Proben die HCV-RNA erfolgreich quantifiziert werden (Range im gepaarten Serum: 477-25.000.000 U/ml; Median: 298.000 U/ml, IQR: 25.050-1.332.500 U/ml). Zwischen DBS- und Serum-HCV-RNA-Werten fand sich eine gute Korrelation (r2=0.94) (vgl. Abbildung).
Für 62 Patienten mit chronischer Hepatitits C wurden die HCV-RNA-Werte in den DBS-Proben mit denen in den gepaarten Serum-Proben verglichen. In 60/62 (97%) der DBS-Proben war HCV-RNA nachweisbar. Im entsprechenden Serum der zwei HCV-RNA-negativen DBS-Proben wurde eine niedrige Viruslast von nur 178 bzw. 331 U/ml gemessen. Fünf DBS-Proben waren zwar HCV-RNA-positiv, jedoch mit Werten unterhalb der Nachweisgrenze von 15 U/ml (HCV-RNA in den dazugehörigen Serumproben: 334 bis 1672 U/ml). Somit konnte in 55/62 (89%) der DBS-Proben die HCV-RNA erfolgreich quantifiziert werden (Range im gepaarten Serum: 477-25.000.000 U/ml; Median: 298.000 U/ml, IQR: 25.050-1.332.500 U/ml). Zwischen DBS- und Serum-HCV-RNA-Werten fand sich eine gute Korrelation (r2=0.94) (vgl. Abbildung).
 Die HCV-RNA-Werten im DBS lagen durchschnittlich 2,27 log U/ml (+/-0,47) tiefer als im Serum, und es zeigte sich eine lineare Beziehung. Nach Anwendung der Korrekturformel log HCV-RNA DBS (korrigiert) = 1,426 + 1,208 x log HCV-RNA DBS war die Korrelation zum Wert im Serum (log HCV-RNA Serum) nahezu 1:1.
Die HCV-RNA-Werten im DBS lagen durchschnittlich 2,27 log U/ml (+/-0,47) tiefer als im Serum, und es zeigte sich eine lineare Beziehung. Nach Anwendung der Korrekturformel log HCV-RNA DBS (korrigiert) = 1,426 + 1,208 x log HCV-RNA DBS war die Korrelation zum Wert im Serum (log HCV-RNA Serum) nahezu 1:1.
4) HCV-Genotypisierung
In 14 zufällig ausgewählten HCV-RNA-positiven Patienten (Range HCV-RNA in den entsprechenden Serum-Proben: 200-1.510.000 U/ml; Median: 337.000 U/ml). Genotyp (1-4) und Subtyp (a-d) stimmten zwischen DBS- und Serumprobe zu 100% überein (5x Genotyp 1, 3x Genotyp 2, 3x Genotyp 3, 3x Genotyp 4).
Conclusion:
- Die HCV-Antikörper-Bestimmung sowie die Quantifizierung und Genotypisierung von HCV-RNA in auf Filterpapier getrocknetem kapillären Vollblut liefert mit kommerziell erhältlichen Assays zuverlässige Ergebnisse (99% Sensitivität und Spezifität der HCV-Antikörper-Bestimmung, HCV-Immunoblot zur Bestätigung aus gleicher Probe möglich, 97% qualitativer und 89% quantifizierbarer HCV-RNA-Nachweis bei im Serum nachweisbarer HCV-RNA, 100% Konkordanz zwischen Genotypisierung im DBS (dried blood spot) und dazugehörigen Serum) und ist somit eine Alternative zur Serum-Analyse.
- Die Vorteile dieser Methode (keine venöse Blutentnahme, vor Ort keine Zentrifuge nötig, Blutproben auf Filterpapier 48h bei Raumtemperatur lager- und verschickbar, danach Lagerung bei -20°C) könnten i.v.-Drogenkonsumenten mit schwierigen Venenverhältnissen den Zugang zu Hepatitis C-Diagnose und -Therapie erleichtern.
- Offen bleibt, ob sich das Verfahren bei einer Sensitivitätsgrenze von ca.1000 U/ml auch für das HCV-RNA-Monitoring unter Therapie eignet.
Quelle: Tuaillon et al., Hepatology 2010;51:752-758